News
新闻动态
探寻AI训练数据源头:生物药物突变库来助力!
“Garbage in,Garbage out”,高质量的数据一直都被认为是AI模型训练的基础。那什么是“高质量的数据”呢?这些数据又是怎么获得的呢?今天我们为大家介绍1篇在2021年由苏黎世联邦理工学院的Sai T Reddy团队发表于Nature Biomedical Engineering的文章:“Optimization of therapeutic antibodies by predicting antigen specificity from antibody sequence via deep learning”。文章表明:通过生物药物突变库筛选获得的生物标签数据是非常适合AI训练的高质量数据。后来文章的作者:Derek M. Mason、Simon Friedensohn、Cédric R. Weber加入了deepCDR Biologics,聚焦AI技术在抗体优化的应用。
前言
目前,抗体的多参数优化非常具有挑战性,主要包括表达水平、粘度、药动学、溶解度和免疫原性等方面,这也使得我们需要花费大量的时间和成本去进行抗体的优化和筛选。2021年4月,苏黎世联邦理工学院的Sai T Reddy团队发表的该文章,利用深度学习在哺乳动物细胞中进行治疗性抗体(全长IgG)的高通量优化筛选(图1)。作者利用表达治疗性抗体曲妥珠单抗(Herceptin)的哺乳动物展示细胞系,结合CRISPR-Cas9介导的同源定向修复(HDR),在变异重链互补决定区3(CDRH3)中引入定点突变库,通过DMS设计筛选出是否对人表皮生长因子受体2(HER2)具有结合性。再利用深度学习预测HER2的特异性,从而筛选出优化后的首选候选药物。
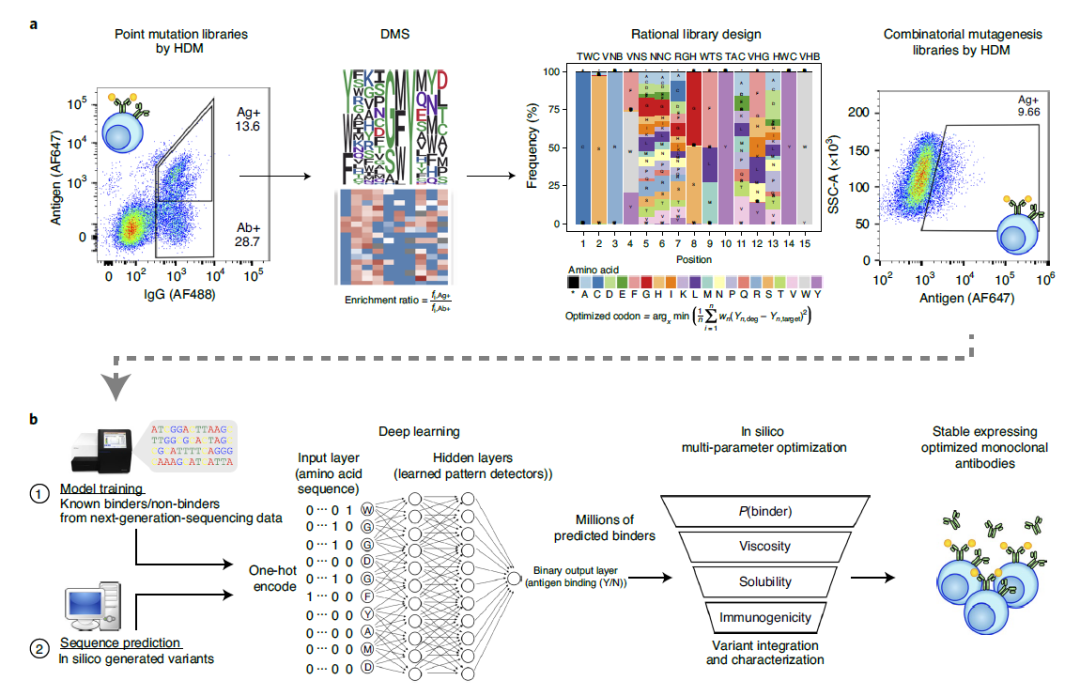
图1.利用深度学习预测抗体靶点特异性
方法和结果
基于CRISPR-Cas9介导的同源定向突变进行DMS。
抗体CDRH3的氨基酸序列是抗原特异性的关键决定性因素,作者通过转染对CDRH3的gRNA和包含在单链寡核苷酸(ssODNs)中的具有NNK简并密码子的组合模板池生成了突变库。然后通过FACS筛选和表达表面IgG的人群,并进行深度测序,且计算出氨基酸的富集率(图2)。利用CRISPR-Cas9介导的同源定向突变在不能结合HER2抗原的曲妥珠单抗变体的混合瘤细胞中生成包含CDRH3变体的库。

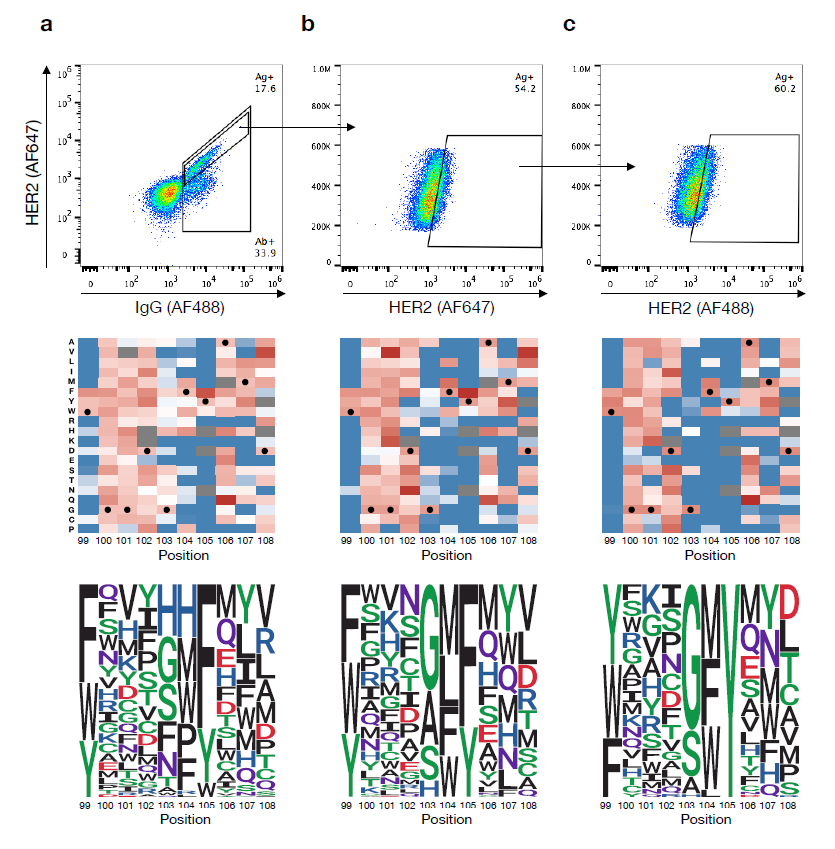
图2.基于CRISPR-Cas9介导的同源定向突变进行DMS
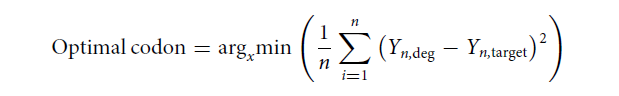
Yn,target为深度测序得到的特定位点特定氨基酸频率,为特定位点给定的简并密码子组编码特定氨基酸的频率,n为氨基酸种类,即20。
组合库的理论蛋白质序列空间为7.17×10^8,远远大于单位点DMS库的多样性,作者通过两轮FACS富集分离抗原结合细胞,并对抗原结合和非抗原结合细胞群体进行深度测序。在组合库中测序数据分别鉴定出11,300个结合体和27,539个非结合体,这些序列变异仅占组合突变库理论蛋白质序列空间的0.0054%(图3)。
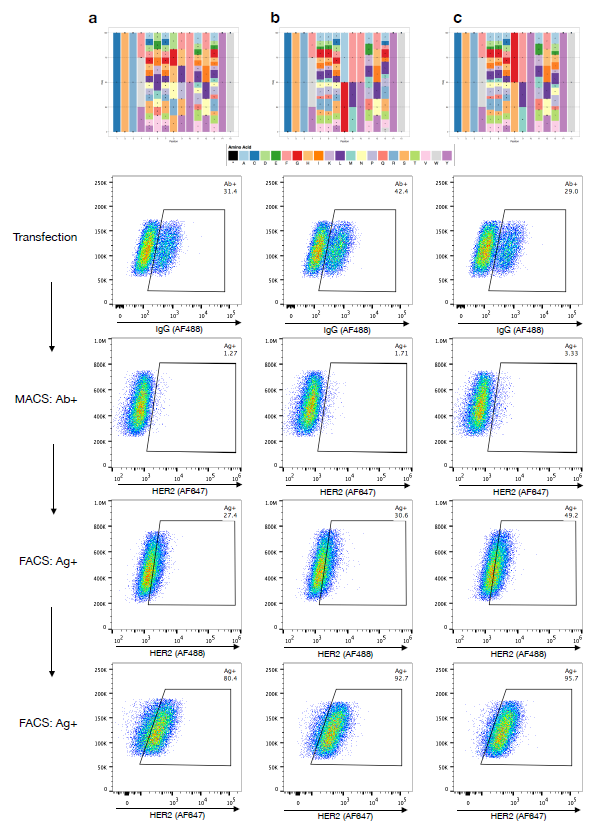
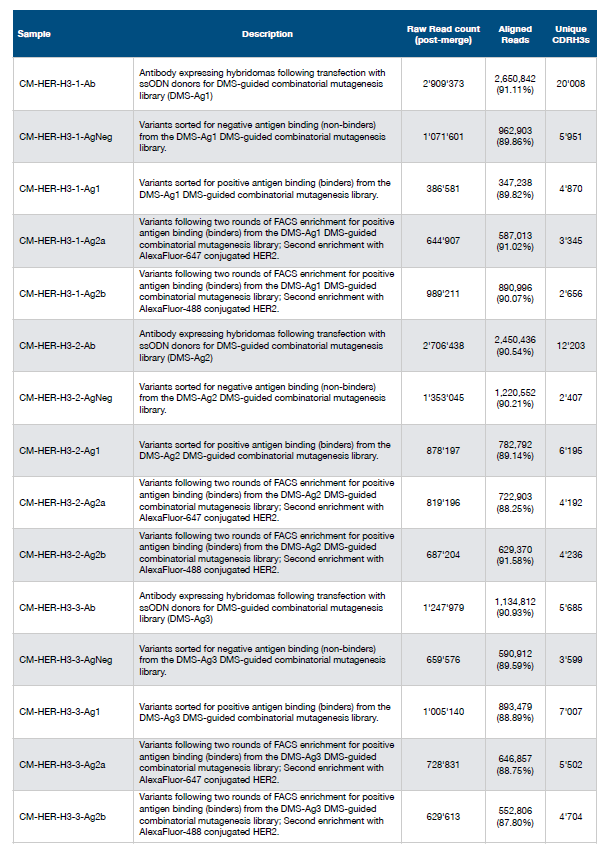
图3.基于序列的突变分析
基于序列的机器学习和深度学习模型,预测抗体特异性。
作者着手开发和训练基于序列的机器学习和深度学习模型,将结合和非结合的CDRH3变体的深度测序数据破译后,通过单热编码将氨基酸序列转换为输入矩阵。前期,作者调查了一系列模型,从现有的测序数据中评估它们在分类结合体和非结合体方面的准确性和精度:一体和二体逻辑回归、k最近邻、支持向量机(线性和高斯核)、标准人工神经网络、长短期记忆循环神经网络(LSTM-RNN)和卷积神经网络(CNN),发现CNN深度学习模型优于其他测试模型,能够准确地对未见过的测试数据进行分类(图4)。
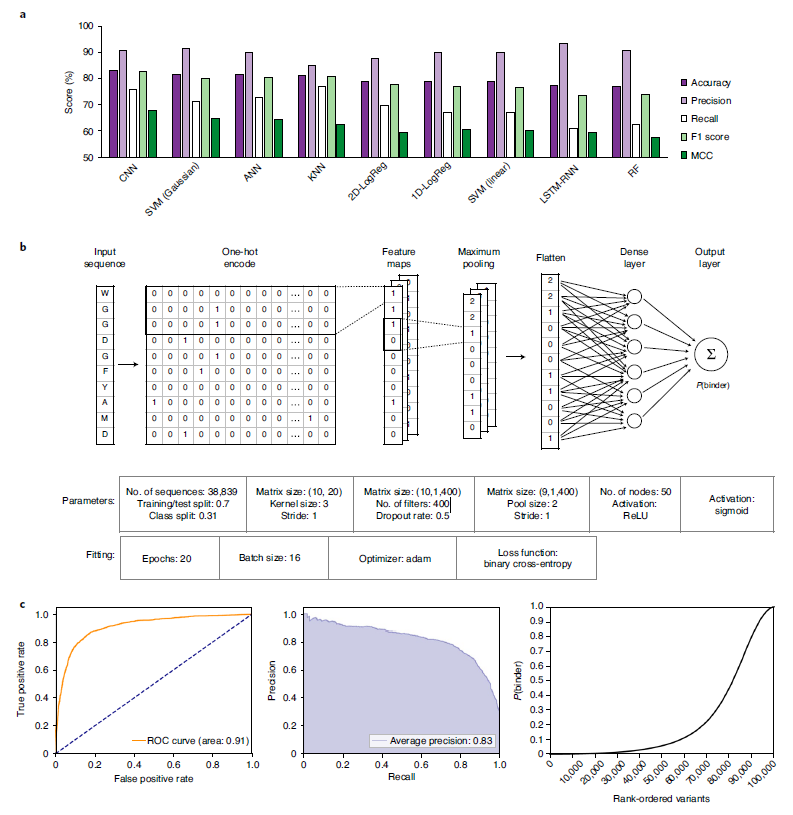
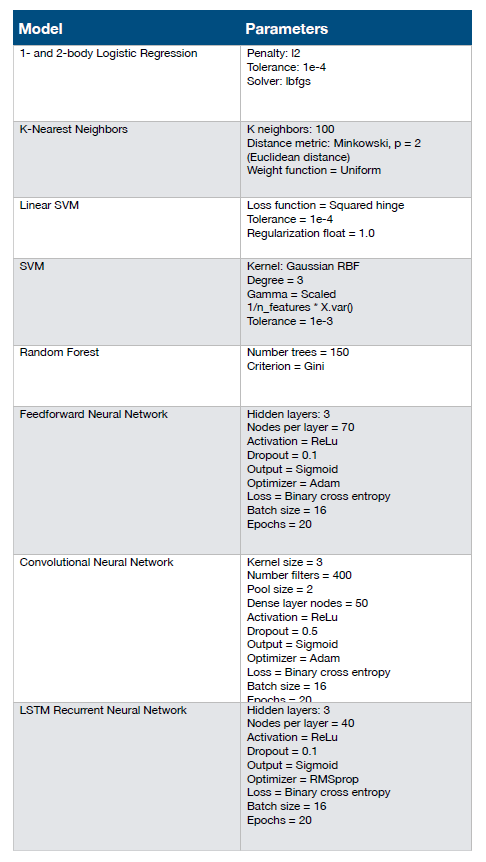
图4.深度学习模型准确预测抗原特异性
作者通过BLI对随机选择的序列进行检测与目标抗原的结合,来评估模型的假阳性和阴性率。发现结合序列都保持了与HER2抗原的显著高亲和性。然而,九个序列中的三个非结合数据也保持了对HER2抗原的亲和性(图5),表明数据集中存在不准确性,这在未来可通过在生成训练数据的FACS期间使用额外的分选策略来解决这个问题。
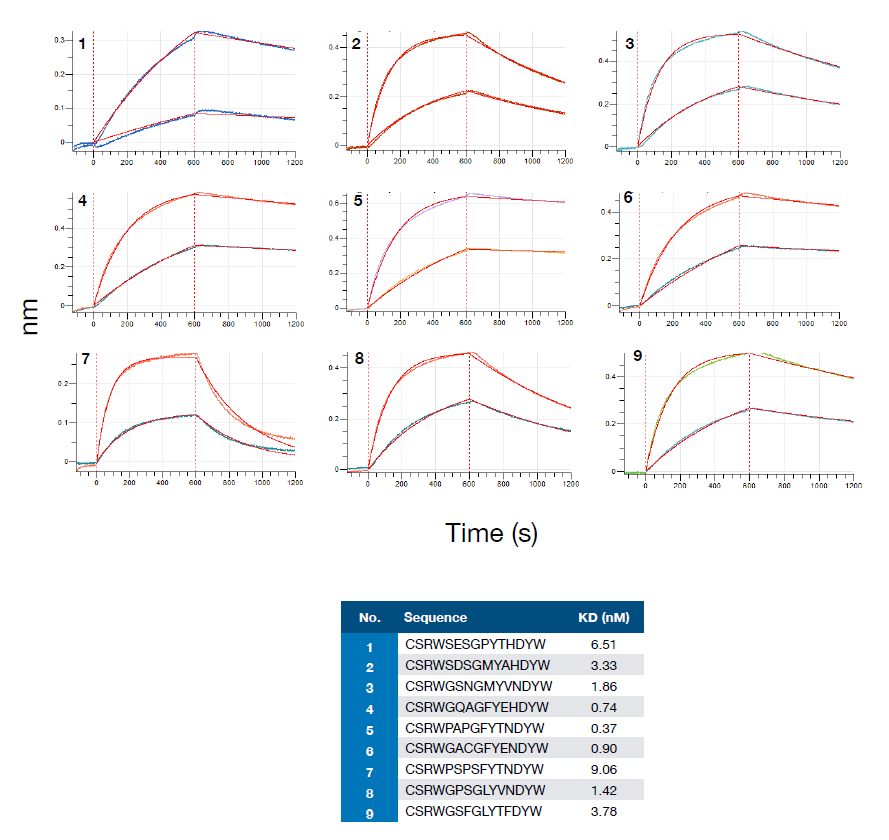

图5.从实验数据集中随机选择序列进行BLI分析
在模型验证的最后一步,作者通过训练神经网络,发现能显示对未见测试数据进行了无差别的序列分类(图6),这表明正确分类的数据训练的网络可以识别学习模式。

图6.模型性能
对选择的序列进行实验表征,筛选出最佳候选序列。
作者利用CRISPR-Cas9 HDR技术生成一个稳定细胞库,进行单细胞分选,并对单克隆变异体做了进一步的表征,最终鉴定到55个突变体。接着使用BLI从细胞上清液中测量它们的表达水平,并显示出不同程度的抗体滴度,其中五个变异体的表达与曲妥珠单抗相当或更好。纯化后,通过荧光测量进行了热稳定性测试,发现10个变异体的热稳定性均与曲妥珠单抗相当或更好(图7)。作者根据使用NetMHCII预测方法预测具有较高免疫原潜力的区域选择了每个变异体和野生型曲妥珠单抗的15个氨基酸片段,发现变异体1和3显示出没有明显的T细胞活化,表明其免疫原性降低。且变体1序列与曲妥珠单抗表达量相当,热稳定性更高,与最初的曲妥珠单抗序列相比,该变体表现出明显的去风险免疫原性潜力。
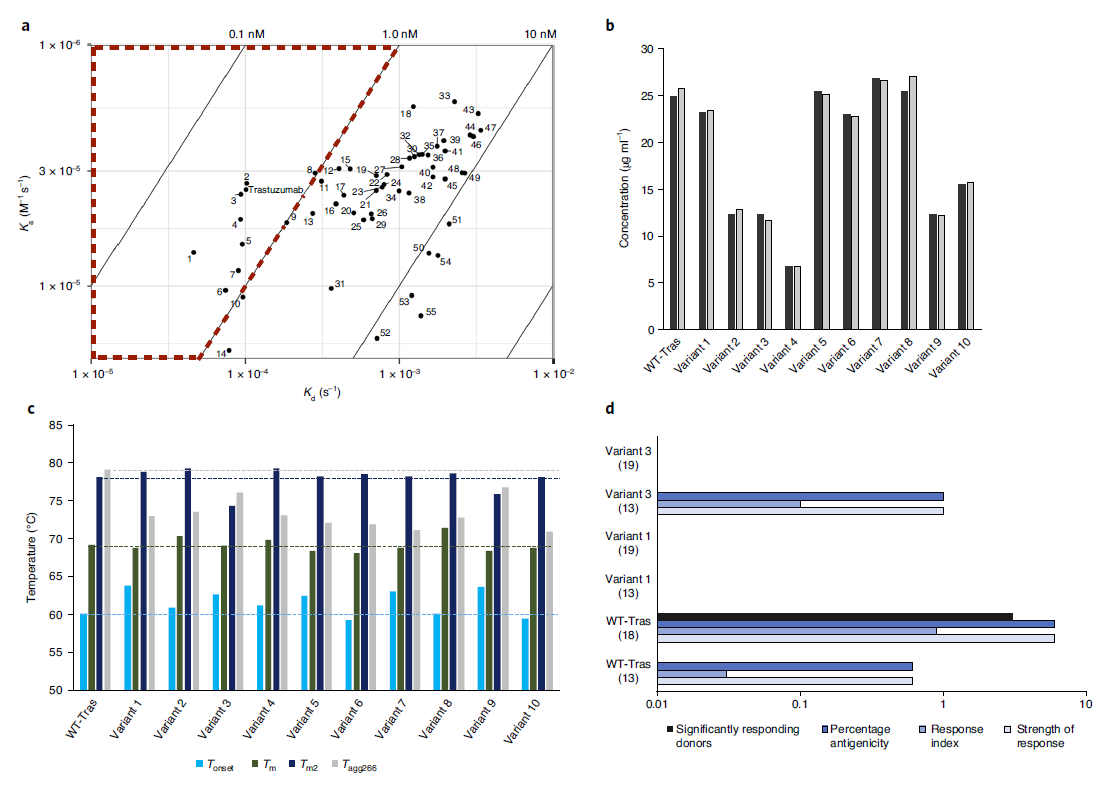
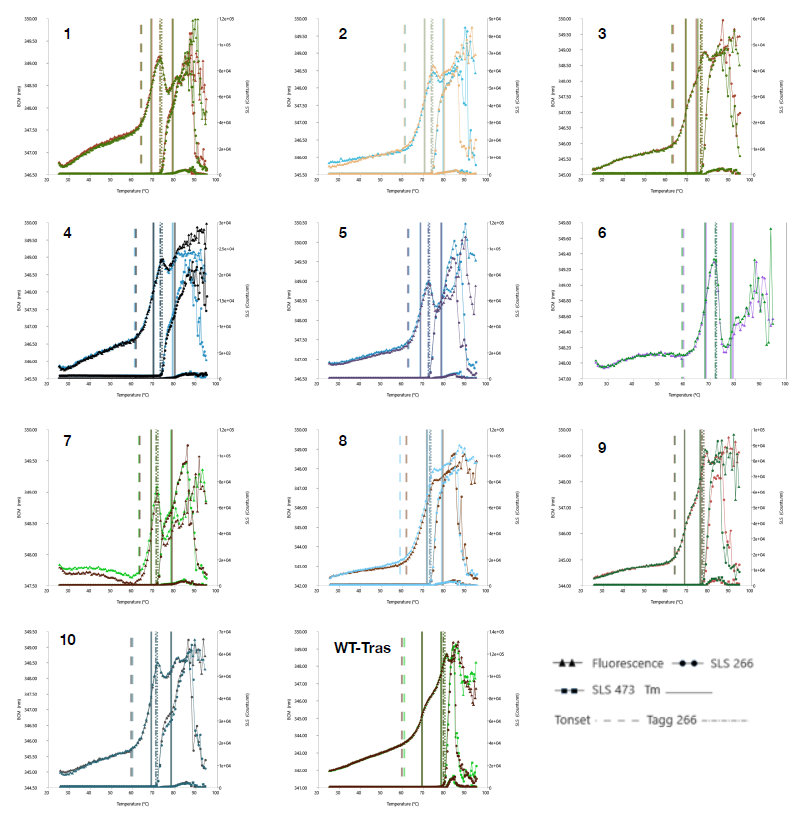
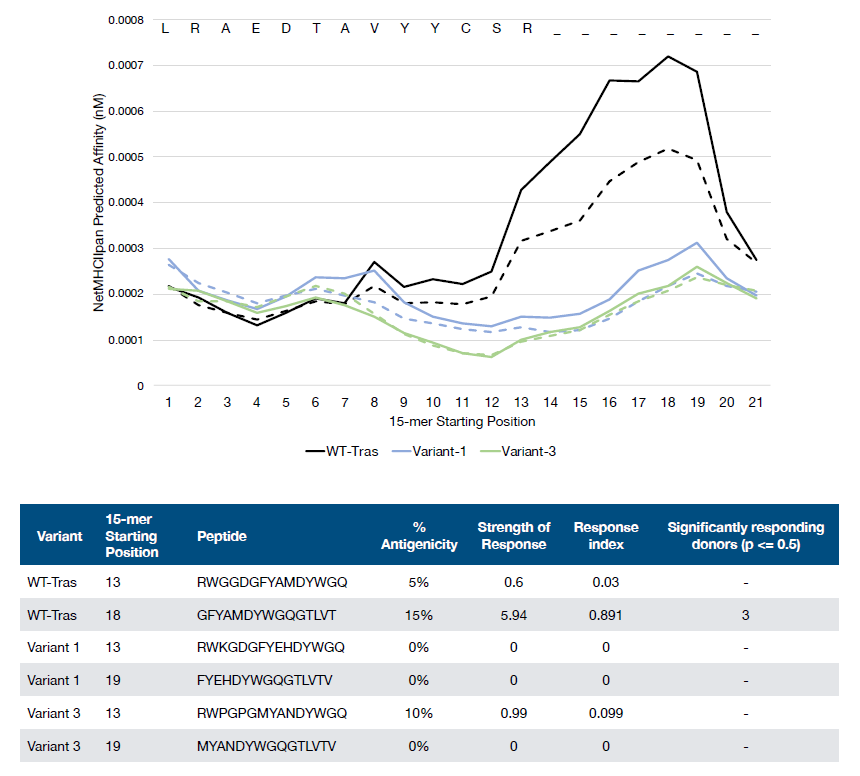
图7.筛选出最佳候选序列
结论
在本文中,作者通过深度学习和抗原特异性检测,获得HER2特异性抗体序列,结合多种生信手段进行目标筛选,得到高度优化的HER2先导序列。本文详细介绍了通过优化基因编辑技术来生成高质量抗体库的方法,所开发的基于深度学习的方法,能够识别高特异性抗原序列,从而大大节省时间和成本,并大幅降低下游临床开发的风险,这项研究推动抗体工程和药物研发具有重要意义,有望为精准医学和个性化治疗的发展提供新的方法和策略。
刘博说:
每一个AI公司都在寻找高质量的数据!
到底什么是高质量数据呢?在本文中,苏黎世联邦理工学院的科学家给出了他们的定义:带标签的(文章中的antigen specificity抗原特异性)、大数据量的(文章中的about 104 variants)、真实筛选获得的(文章中从Combinatorial mutagenesis libraries筛选富集的)的抗体氨基酸序列数据。使用这些高质量数据,作者对多种机器或者深度学习模型进行了训练和评价,并最终确认卷积神经网络(CNN)在准确率(accuracy)、精确率(precision)等多项指标上的表现最为优异,适合用于以氨基酸序列为基础的抗体优化过程。
到底怎么样才能获得高质量数据呢?深度学习(如文章中的CNN)对数据的标签、数量都有很高的要求。但是,从公开数据库获得的数据,标签是相对单一的;而从文献专利获得的数据,是片段化的,标签是无序的;因此这些数据在特征抓取、合并、关联和高维变换上并不方便,在对AI算法模型的训练中有很多的局限。
本文使用的是一种利用抗体突变库技术获得高质量数据的方法:首先通过随机突变或者理性设计突变,使哺乳动物细胞表面展示抗体突变库,通过流式分选并富集与抗原特异性结合的抗体氨基酸序列。通常,从抗体突变库获得的抗体序列会有以下的一些特点:
1)序列是带标签的。本文是基于抗原结合力进行的筛选,所以序列所带的标签为“抗原特异性”。如果我们更换筛选条件,比如基于表达量筛选、基于功能性筛选、或者基于稳定性筛选,那么获得的序列就会带上“表达量”、“激活/阻断能力”、“结构稳定性”等不同的标签。
2)序列是连续的。通过对抗体单个位点或者多个位点进行饱和突变或者设计突变,就能够获得在某个位点或者多个位点的连续的氨基酸变化,帮助AI对这些位点连续的、细微差别的变化进行学习。
3)序列是排序的。如果我们是对抗体的亲和力进行筛选,在对富集的序列进行NGS测序后,可以获得每个抗体序列的数目和频率,理论上数目越多、频率越高,就意味着富集的程度越高、与抗原的亲和力越高,这样就能对NGS获得的序列(通常为几万到几十万条序列,取决于测序的深度)进行相对准确的排序。
4)序列是能够进行阳性/阴性数据分群的,比如文章中的binder和non-binder抗体氨基酸序列。
因此,相对于公开数据库和文献数据,人工突变库筛选产生的数据,精准度和连续性更好,也可以理解为“分辨率”更高。
尽管文章只是在抗原特异性这一标签或者维度开展了AI抗体优化的工作,但却证明了“利用人工突变库生产的标签数据”用于AI训练的可行性和有效性。文中的CRISPR-Cas9突变和哺乳动物细胞展示只是构建人工突变库的一种方式,如果扩展到更多的突变技术和展示技术,比如全合成库技术和噬菌体展示技术,产生的序列数量和序列所带标签的种类将会更多,比如耐高温、pH敏感性、蛋白酶抵抗等各种各样的标签。这些序列数据对于AI的学习训练也会更有帮助。
刘江海
加拿大Saskatchewan大学博士
盛世君联公司创始人、CEO,曾在加拿大萨省治疗性抗体资源中心就读博士后。先后获得四川省“千人计划(创业领军)、“蓉漂人才”计划,和“金熊猫人才”计划。在全合成库技术、生物药物库设计和构建、抗体发现和优化领域具有丰富的实战经验,领导了多个单抗、双抗、CAR-T药物的临床前开发。
